热图是做分析时常用的展示方式,简单、直观、清晰。可以用来显示基因在不同样品中表达的高低、表观修饰水平的高低等。任何一个数值矩阵都可以通过合适的方式用热图展示。
本篇使用r的ggplot2包实现从原始数据读入到热图输出的过程,并在教程结束后提供一份封装好的命令行绘图工具,只需要提供矩阵,即可一键绘图。
上一篇讲述了rstudio的使用作为r写作和编译环境的入门,后面的命令都可以拷贝到rstudio中运行,或写成一个r脚本,使用rscript heatmap.r运行。我们还提供了bash的封装,在不修改r脚本的情况下,改变参数绘制出不同的图形。
绘图首先需要数据。通过生成一堆的向量,转换为矩阵,得到想要的数据。
data <- c(1:6,6:1,6:1,1:6, (6:1)/10,(1:6)/10,(1:6)/10,(6:1)/10,1:6,6:1,6:1,1:6, 6:1,1:6,1:6,6:1) [1] 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0 2.0 1.0 6.0 5.0 4.0 3.0 2.0 1.0 1.0
[20] 2.0 3.0 4.0 5.0 6.0 0.6 0.5 0.4 0.3 0.2 0.1 0.1 0.2 0.3 0.4 0.5 0.6 0.1 0.2
[39] 0.3 0.4 0.5 0.6 0.6 0.5 0.4 0.3 0.2 0.1 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0
[58] 3.0 2.0 1.0 6.0 5.0 4.0 3.0 2.0 1.0 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0
[77] 2.0 1.0 1.0 2.0 3.0 4.0 5.0 6.0 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0 2.0
[96] 1.0注意:运算符的优先级。
> 1:3 4
[1] 5 6 7
> (1:3) 4
[1] 5 6 7
> 1:(3 4)
[1] 1 2 3 4 5 6 7vector转为矩阵 (matrix),再转为数据框 (data.frame)。
# ncol: 指定列数
# byrow: 先按行填充数据
# ?matrix 可查看函数的使用方法
# as.data.frame的as系列是转换用的
data <- as.data.frame(matrix(data, ncol=12, byrow=t))v1 v2 v3 v4 v5 v6 v7 v8 v9 v10 v11 v12
1 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0 2.0 1.0
2 6.0 5.0 4.0 3.0 2.0 1.0 1.0 2.0 3.0 4.0 5.0 6.0
3 0.6 0.5 0.4 0.3 0.2 0.1 0.1 0.2 0.3 0.4 0.5 0.6
4 0.1 0.2 0.3 0.4 0.5 0.6 0.6 0.5 0.4 0.3 0.2 0.1
5 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0 2.0 1.0
6 6.0 5.0 4.0 3.0 2.0 1.0 1.0 2.0 3.0 4.0 5.0 6.0
7 6.0 5.0 4.0 3.0 2.0 1.0 1.0 2.0 3.0 4.0 5.0 6.0
8 1.0 2.0 3.0 4.0 5.0 6.0 6.0 5.0 4.0 3.0 2.0 1.0# 增加列的名字
colnames(data) <- c("zygote","2_cell","4_cell","8_cell","morula","icm","esc","4 week pgc","7 week pgc","10 week pgc","17 week pgc", "oocyte")
# 增加行的名字
# 注意paste和paste0的使用
rownames(data) <- paste("gene", 1:8, sep="_")
# 只显示前6行和前4列
head(data)[,1:4] zygote 2_cell 4_cell 8_cell
gene_1 1.0 2.0 3.0 4.0
gene_2 6.0 5.0 4.0 3.0
gene_3 0.6 0.5 0.4 0.3
gene_4 0.1 0.2 0.3 0.4
gene_5 1.0 2.0 3.0 4.0
gene_6 6.0 5.0 4.0 3.0虽然方法比较繁琐,但一个数值矩阵已经获得了。
还有另外2种获取数值矩阵的方式。
- 读入字符串
# 使用字符串的好处是不需要额外提供
# 简单测试时可使用,写起来不繁琐,又方便重复
# 尤其适用于在线提问时作为测试案例
> <- "id;zygote;2_cell;4_cell;8_cell
gene_1;1;2;3;4
gene_2;6;5;4;5
gene_3;0.6;0.5;0.4;0.4"
# 习惯设置quote为空,避免部分基因名字或注释中存在引号,导致读入文件错误。
# 具体错误可查看 http://blog.genesino.com/collections/r_tips/ 中的记录
> data2 <- read.table(text=txt,sep=";", header=t, row.names=1, quote="")
> head(data2)
zygote x2_cell x4_cell x8_cell
gene_1 1.0 2.0 3.0 4.0
gene_2 6.0 5.0 4.0 5.0
gene_3 0.6 0.5 0.4 0.4可以看到列名字中以数字开头的列都加了x。一般要尽量避免行或列名字以数字开头,会给后续分析带去一些困难;另外名字中出现的非字母、数字、下划线、点的字符都会被转为点,也需要注意,尽量只用字母、下划线和数字。
# 读入时,增加一个参数`check.names=f`也可以解决问题。
# 这次数字前没有再加 x 了
> data2 <- read.table(text=txt,sep=";", header=t, row.names=1, quote="", check.names = f)
> head(data2)
zygote 2_cell 4_cell 8_cell
gene_1 1.0 2.0 3.0 4.0
gene_2 6.0 5.0 4.0 5.0
gene_3 0.6 0.5 0.4 0.4- 读入
与上一步类似,只是改为文件名,不再赘述。
> data2 <- read.table("filename",sep=";", header=t, row.names=1, quote="")数据读入后,还需要一步格式转换。在使用ggplot2作图时,有一种长表格模式是最为常用的,尤其是数据不规则时,更应该使用 (这点,我们在讲解箱线图时再说)。
# 如果包没有安装,运行下面一句,安装包
#install.packages(c("reshape2","ggplot2"))
library(reshape2)
library(ggplot2)
# 转换前,先增加一列id列,保存行名字
data$id <- rownames(data)
# melt:把正常矩阵转换为长表格模式的函数。工作原理是把全部的非id列的数值列转为1列,命名为value;所有字符列转为variable列。
# id.vars 列用于指定哪些列为id列;这些列不会被merge,会保留为完整一列。
data_m <- melt(data, id.vars=c("id"))
head(data_m) id variable value
1 gene_1 zygote 1.0
2 gene_2 zygote 6.0
3 gene_3 zygote 0.6
4 gene_4 zygote 0.1
5 gene_5 zygote 1.0
6 gene_6 zygote 6.0
7 gene_7 zygote 6.0
8 gene_8 zygote 1.0
9 gene_1 2_cell 2.0
10 gene_2 2_cell 5.0
11 gene_3 2_cell 0.5
12 gene_4 2_cell 0.2
13 gene_5 2_cell 2.0
14 gene_6 2_cell 5.0
15 gene_7 2_cell 5.0
16 gene_8 2_cell 2.0数据转换后就可以画图了,分解命令如下:
# data_m: 是前面费了九牛二虎之力得到的数据表
# aes: aesthetic的缩写,一般指定整体的x轴、y轴、颜色、形状、大小等。
# 在最开始读入数据时,一般只指定x和y,其它后续指定
p <- ggplot(data_m, aes(x=variable,y=id))
# 热图就是一堆方块根据其值赋予不同的颜色,所以这里使用fill=value, 用数值做填充色。
p <- p geom_tile(aes(fill=value))
# ggplot2为图层绘制,一层层添加,存储在p中,在输出p的内容时才会出图。
p
## 如果你没有使用rstudio或其它r图形版工具,而是在远程登录的服务器上运行的交互式r,需要输入下面的语句,获得输出图形 (图形存储于r的工作目录下的rplots.pdf文件中)。
## 如何指定输出,后面会讲到。
#dev.off()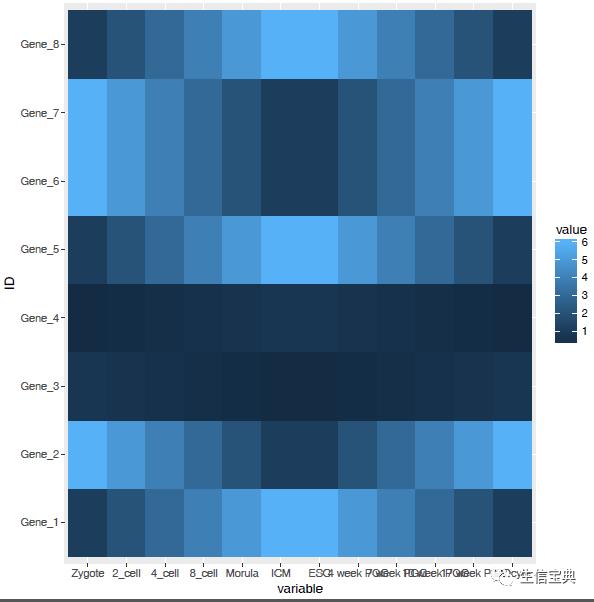
热图出来了,但有点不对劲,横轴重叠一起了。一个办法是调整图像的宽度,另一个是旋转横轴标记。
# theme: 是处理图美观的一个函数,可以调整横纵轴label的选择、图例的位置等。
# 这里选择x轴标签45度。
# hjust和vjust调整标签的相对位置,具体见 。
# 简单说,hjust是水平的对齐方式,0为左,1为右,0.5居中,0-1之间可以取任意值。vjust是垂直对齐方式,0底对齐,1为顶对齐,0.5居中,0-1之间可以取任意值。
p <- p theme(axis.text.x=element_text(angle=45,hjust=1, vjust=1))
p 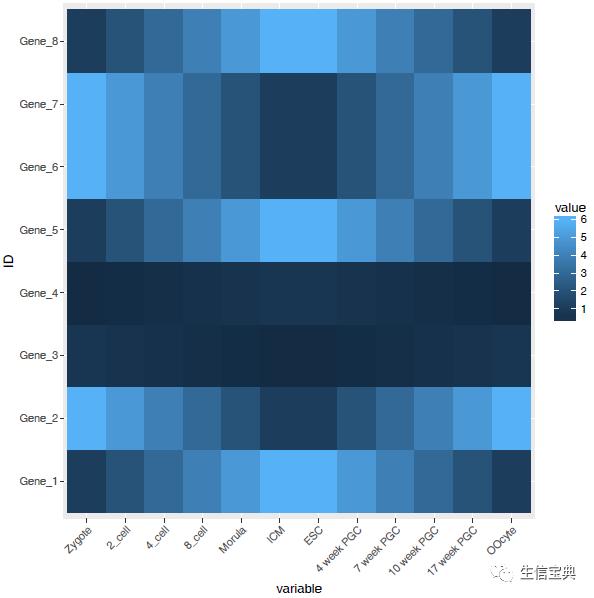
设置想要的颜色。
# 连续的数字,指定最小数值代表的颜色和最大数值赋予的颜色
# 注意fill和color的区别,fill是填充,color只针对边缘
p <- p scale_fill_gradient(low = "white", high = "red")
p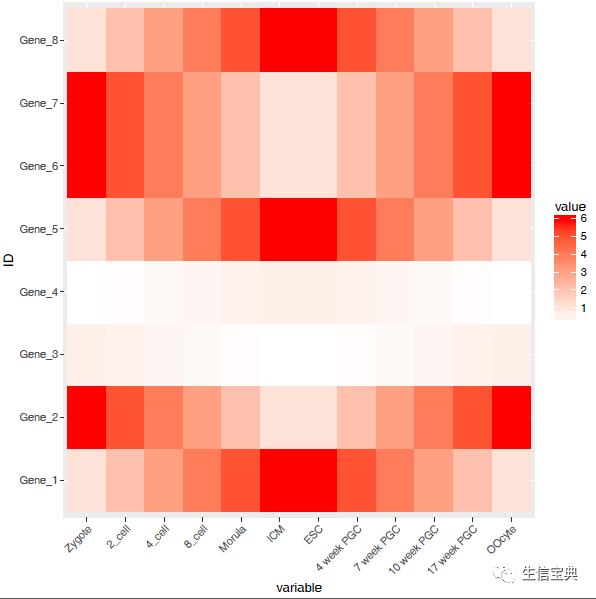
调整legend的位置。
# postion可以接受的值有 top, bottom, left, right, 和一个坐标 c(0.05,0.8) (左上角,坐标是相对于图的左下角计算的)
p <- p theme(legend.position="top")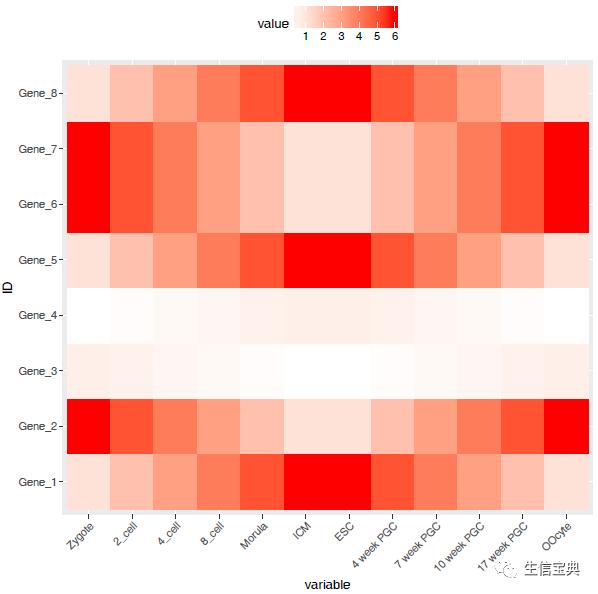
调整背景和背景格线以及x轴、y轴的标题。(注意灰色的背景没了)
p <- p xlab("samples") theme_bw() theme(panel.grid.major = element_blank()) theme(legend.key=element_blank())
p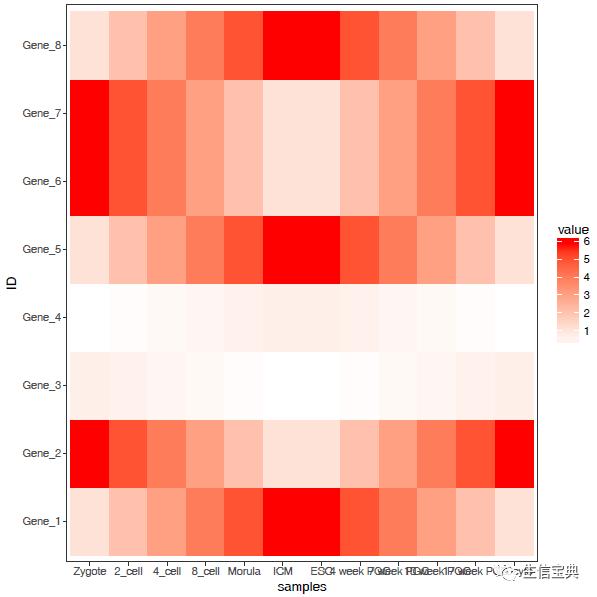
合并以上命令,就得到了下面这个看似复杂的绘图命令。
p <- ggplot(data_m, aes(x=variable,y=id)) xlab("samples") theme_bw() theme(panel.grid.major = element_blank()) theme(legend.key=element_blank()) theme(axis.text.x=element_text(angle=45,hjust=1, vjust=1)) theme(legend.position="top") geom_tile(aes(fill=value)) scale_fill_gradient(low = "white", high = "red")图形出来了,就得考虑存储了,
# 可以跟输出文件不同的后缀,以获得不同的输出格式
# colormode支持srgb (屏幕)和cmyk (打印,部分杂志需要,看上去有点褪色的感觉)格式
ggsave(p, filename="heatmap.pdf", width=10,
height=15, units=c("cm"),colormodel="srgb")至此,完成了简单的heatmap的绘图。但实际绘制时,经常会碰到由于数值变化很大,导致颜色过于集中,使得图的可读性下降很多。因此需要对数据进行一些处理,具体的下次再说。
如若转载,请注明出处:https://www.dasum.com/107871.html